MestReNova(Mnova)14中文破解版是专业的核磁共振和LC/GC/MS数据处理、可视化、模拟、预测、演示和分析软件包,Mnova的设计旨在使NMR和LC/GC/MS更加便捷和强大,并以最小的努力为用户提供更好的结果。使用旨在让那您能够快速通过使用它来获得良好的效果,具有完整的功
MestReNova(Mnova)14中文破解版是专业的核磁共振和LC/GC/MS数据处理、可视化、模拟、预测、演示和分析软件包,Mnova的设计旨在使NMR和LC/GC/MS更加便捷和强大,并以最小的努力为用户提供更好的结果。使用旨在让那您能够快速通过使用它来获得良好的效果,具有完整的功能和领先的技术,带来全新的概念和发展,不仅包括MestReC中存在的所有NMR处理和分析功能,并且能够在组织内部存储和共享数据,改变科学家处理和分析数据的方式时开辟全新的可能性。报告NMR和LC/GC/MS数据。此外,该程序为大多数NMR和LC/GC/MS光谱仪和色谱图格式提供了多种转换工具,并包括NMR程序的所有常规处理,显示和绘图功能,以及更先进的处理技术以及非常友好的用户界面图形界面。全新破解版下载,含破解补丁文件,有需要的朋友不要错过了!

安装激活教程
1、在本站下载并解压,如图所示

2、双击MestReNova-14.2.0-26256_x64.msi运行安装,勾选我接受许可证协议条款

3、选择软件安装路径

4、安装完成,退出向导
5、将crack中的文件复制到安装目录中,点击替换目标中的文件

6、运行后,点击编辑,选择高级插件来安装高级插件
MestReNova v14.2新功能
1、核磁共振
记住布局模板中的峰/多峰报告选项
提高2D NMR的显示阈值
能够定义频率偏移(ppm)
能够更改2D堆栈中单个光谱的颜色
修复qGSD负峰
所有实验轴的配置
仅在施加阶数为1的相位值时才以阶数0进行自动相位校正
添加新选项(“属性”对话框)以修改“多重框”属性
NMR CSV矩阵涉及盲区
堆栈自动缩放现在考虑了盲区或割伤
更好的自动阈值显示2D NMR
支持1D Q.Mnova中的一种文件格式
改善2D峰和2D多重峰属性以进行可视化
导入TopSpin峰列表
更改默认2D调色板的功能
多重报告:用于选择哮喘类型的新选项
分配方面的改进:
现在分配峰更容易
将核符号添加到2D分配标签
分配问题2D聚类(多重)
2D多重峰的分配质量
作业中的背景多重峰:1H和其他2D光谱不同于HSQC
将“不稳定”和“质量”属性添加到MolecularAssignmentsParser
改善一维和二维光谱中的突出显示
2、Mnova MS
能够打开Shimadzu GC/MS数据
色谱减法获得的改进结果
在所有可用的MS²光谱上方显示概览表
通过脚本对LCMS跟踪进行自动基线校正
可以将结构分配给TIC(或其他迹线)中的峰
新的十字准线模式:MS光谱提取解卷积
能够“手动创建”多个质量范围的色谱图
计算相对质量的元素组成的能力
能够复制和粘贴单个图并保持其大小
能够显示来自多个测量文件的多个光谱
新进口商开放JEOL Accu-ToF 4G LC-Plus
在同一窗口的TIC(色谱图重叠图)下方显示的EIC
在不删除或覆盖文档先前分析的情况下,将布局模板应用于MS数据的选项
将布鲁克Compass Xtract Windows二进制文件捆绑到Mnova安装程序中
3、Mnova NMRPredict桌面
预测并突出显示改进
通过类似Karplus的方程式(例如Altona)计算3J[HCCH]值
咪唑衍生物的4J(HH)预测
4、Mnova GUI
可以将ID分配给布局模板对象
布局模板(分子)
报告正确数量的参数
改善MNova中的报告功能
堆叠NMR光谱的用户友好选择
在“拆分文档”脚本中添加“全部应用于”选项
能够导入前缀为UTF-8 BOM的JCAMP-DX文件
为Mnova实施循环日志
添加快捷方式以激活键盘分子编辑器模式
在Mnova的NMR Tools菜单中添加“合成纯位移”脚本
SQA报告峰高
包括导出/导入NMReDATA文件时的处理
可以直接从.sdf文件(NMReData)导入NMR数据的功能
5、Mnova数据库
改进保存数据库浏览器的新首选项时获得的消息
根据上下文使用不同的DBBrowser布局
数据库文本搜索的完成程序应区分大小写
在搜索结果中对齐质谱图
DBrowser工具栏的改进
更新dbPeakSearch示例脚本
默认情况下能够通过降低分数显示搜索结果
6、Mnova ElViS
实现Thermo Scientific/Nicolet OMNIC文件格式的转换器
支持SP格式
为序列化对象的功能metaData添加ElViS支持
能够读取B&WTek编码的SPC文件
7、分子图
通用结构的新模板
允许用户创建自己的模板
绘制亲缘关系的能力
绘制激进和孤对的能力
碎片支持
仅使用键盘快捷键即可绘制分子
将双键当量(DBE)添加到化合物表中的分子特性中
计算3D分子中显性质子的正确3D坐标
J耦合的色图
使用上下文菜单添加更改键类型的功能
在公式工具提示中添加下标数字。
“分子模板”表中的改进
检测立体时要考虑原子同位素
重新设计分子功能区界面
8、IUPAC名称
使用IUPAC名称工具后对原子重新编号-这样两个编号系统是一致的
带主电荷的化合物的命名(过氧硫酸酯和硫代过氧酸酯)
用自由基命名分子
在未扩展标签的结构中使用“生成IUPAC编号”
列举无机分子的易例
用同位素命名分子
9、Mnova脚本
可从脚本引擎作为默认值访问质量分子匹配选项
可以从脚本引擎访问质量分子匹配的默认值
改善脚本引擎的按钮图标配置
NMRProcessing的更新文档
通过脚本中的多个操作启用按钮的创建
可从脚本访问分子匹配中使用的公式
能够将xml svg加载到Pixmap中
创建一个类似于QColorDialog的QFontDialog包装器
获取标签的标签文本应可从脚本引擎访问
在脚本中访问创建具有线条和散点图的类所需的类
10、Mnova装订
合并的新报告
选择在批处理期间关闭文档。
11、结构阐明
处理分解碳信号的能力
结构生成器。允许生成具有63个以上原子的结构。
12、化学计量学
在PCA中的2D分数图中添加置信椭圆
创建PCA测试工作流程
PCA功能应提供距离计算
Biologics HOS 1D PROFILE分析方法
Bin归一化可以应用于指定的光谱区域
导出/导入Bin矩阵的能力
ECHOS和CCSD分析应允许用户指定标题
ECHOS矩阵分析
CCSD矩阵分析
Bin宽度应允许4个十进制数字
BioHOS(工作区)结果报告功能
保存/加载PCA模型的能力
设置PCA元数据的能力
PCA:改进为得分和负荷图选择组件的方法
PCA模型的交叉验证(CV)
在PCA分析之后导出特征值/特征向量
改善ECHOS中的绘图性能
在“ECHOS选项”对话框中添加一条信息消息,以了解在“自动”模式下对光谱所做的更改
一维分析前从蛋白质光谱中去除赋形剂和缓冲液信号
一维轮廓:建议处理
使标题列在CCSD结果的摘要表中可调整大小
矩阵评估表标题中的更好工具提示
·...和更多的小错误已修复。
我们的计划是大约每四个月分发一个新版本。在此期间,您可以期望看到添加到软件中的功能:
·光谱的导数处理
·GEN-2D NMR,用于提高间接尺寸分辨率的处理。
·光谱编辑
·图形用户界面增强
·新的分析工具
我们希望您会发现此版本的Mnova是市场上最完整的NMR处理和分析工具之一,并结合了革命性的界面和许多新颖,原创和创新的功能。
使用帮助
快速可视化指南,用于处理常规1D-NMR数据集
您可以按照菜单“文件/帮助/下载示例数据集”找到奎宁数据集。
请按照以下步骤处理常规1D-NMR光谱:
1、将包含FID文件的文件夹拖放到Mnova中,以完全处理光谱。如果该文件夹包含.mol文件,则还将显示分子结构。(您也可以按照菜单“文件/打开”)
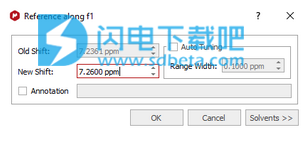
2、如果未参考光谱,则可以通过单击“参考”按钮(在分析功能区下方)TMS进行校准,然后选择要作为参考点的峰。
3、单击“基线校正”图标基线图标,以校正光谱的基线。
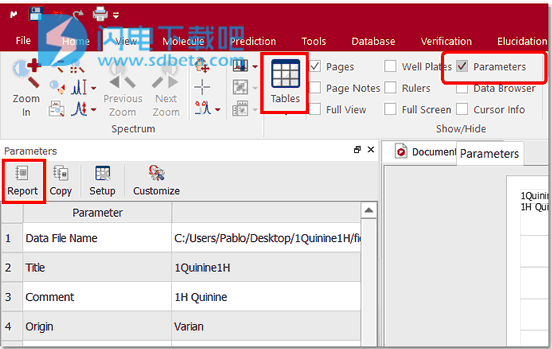
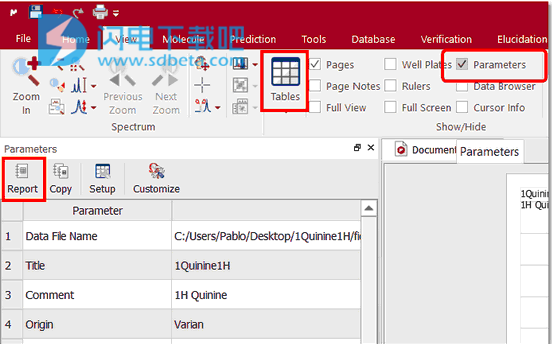
4、要将参数表粘贴在光谱窗口上,只需遵循菜单:“视图/表/参数”,然后单击“报告”图标。请记住,您只需单击并拖动任何边框即可调整任何对象的大小。在这种情况下,频谱已调整大小,以便将表格放在页面的右侧。
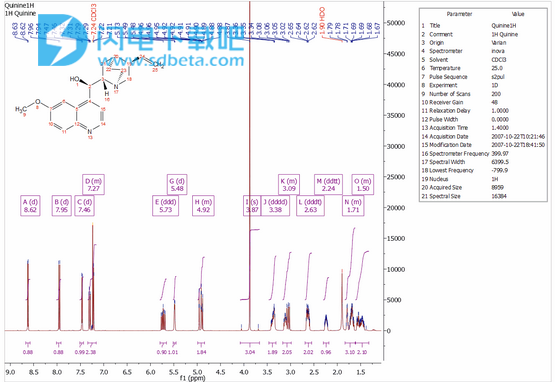
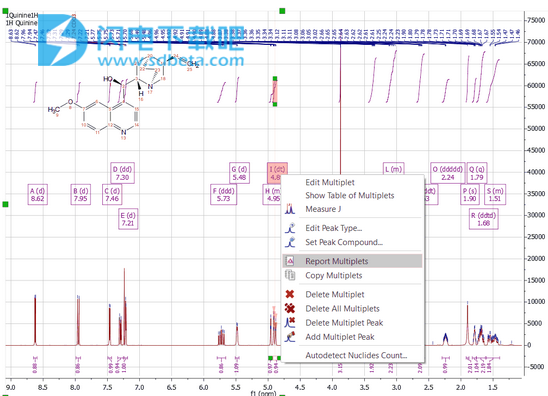
5、单击“Multiplet Analysis”图标“Multiplet”图标,应用自动多重分析。它将应用具有溶剂和杂质识别功能的自动峰采选分析。如您在下面的示例中看到的,已检测到CDCl3信号,并且其峰值标签显示为红色。
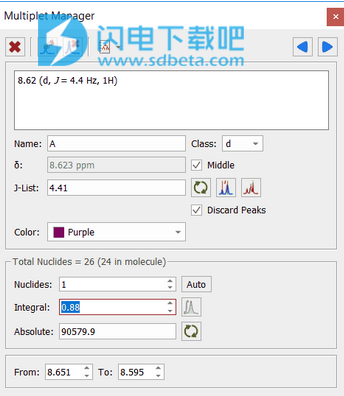
6、双击多重框以标准化“多重积分”值:
7、要获取多重报告列表,只需右键单击多重框并选择“报告多重”:
请记住,您只需单击并拖动即可自由移动任何Mnova对象。在上面的示例中,我们移动了分子结构和多重峰报告
8、您可以通过以下方式将多重报告复制到剪贴板:右键单击多重框中并选择“复制多重”(或通过在多重表上选择“复制文本”),然后仅通过使用Ctrl+将报告粘贴到另一个文档中V(在Mac中为Cmd+V)。
9、最后,通过单击“文件功能区”下(或工具栏上)的相应图标,您可以将文档保存,打印或导出为PDF:
二、快速视觉指南,处理常规2D-NMR数据集
(在Mnova Lite中不可用)
您可以按照菜单“文件/帮助/下载示例数据集”找到奎宁数据集。
处理2D-NMR光谱的过程与一维光谱非常相似:
1、拖放包含光谱文件的文件夹,或单击“文件/打开”按钮打开图标,然后选择FID文件(或Bruker中的SER文件)以获得已处理的2D-NMR光谱。Mnova将为您选择处理功能(FT,相位校正等),但是,当然,您可以更改和优化其中的任何功能,然后根据需要重新处理数据。
2、如果在“文件/首选项/NMR/导入”菜单下选中了适用的选项,则Mnova自动检测一维光谱并将其用作2D图的外部迹线。
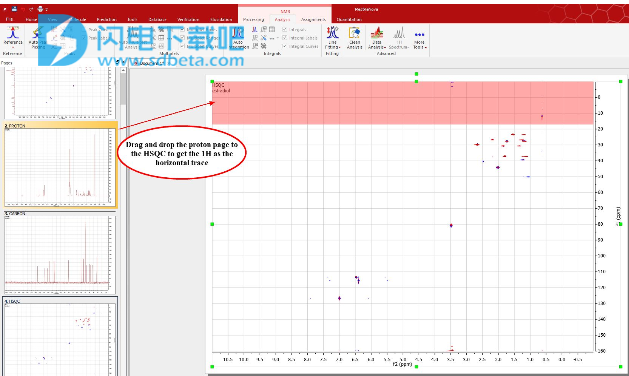
如果未选中“自动附加轨迹”选项,则可以将任何当前打开的1D-NMR光谱拖放到2D NMR数据集中,以将其作为轨迹。在这里,您可以看到如何将包含1H数据集的页码1放到HSQC上以获得水平轨迹:
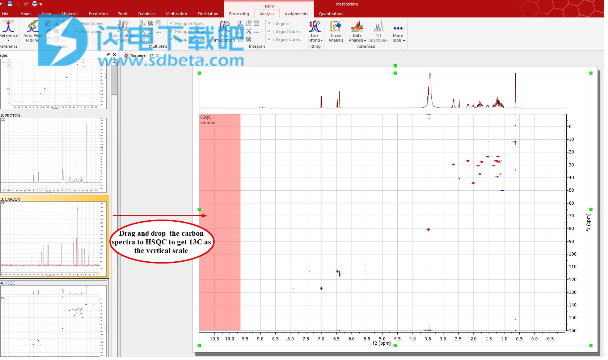
可以对第2页(包含13C光谱)执行相同操作以获得垂直轨迹:
在这里您可以看到结果:
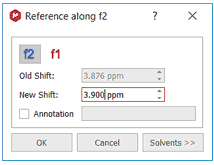
3、要校准光谱,只需单击“参考”工具栏上的TMS按钮,然后选择要作为参考点的峰。最后,沿f2然后沿f1选择所需的参考:
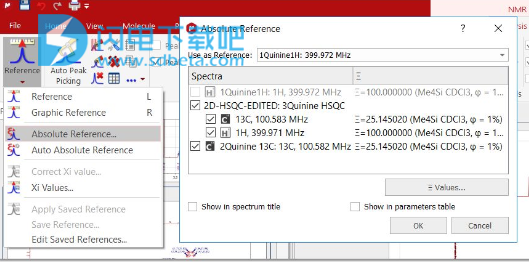
如果您已加载1H和13C NMR光谱,则可以通过单击工具栏上的“绝对参考”按钮(或按照菜单“分析/参考”)使用“绝对参考”功能来参考2D和C-13。/绝对参考'):
4、单击“Peak Picking”图标“Peak”图标以运行“Peak Picking”分析,并按照菜单“View/Tables/Parameters”显示“Parameters Table”。
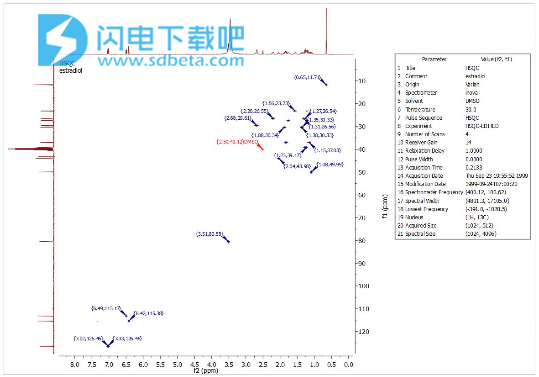
5、然后,单击“报告”按钮以将表格粘贴到页面上,并将其向右移动(通过单击和拖动)。最后调整2D光谱的大小以获取以下报告:
 化学分析数据处理套件 Mestrelab MestReNova v14.2.0.26256 中文激活版
化学分析数据处理套件 Mestrelab MestReNova v14.2.0.26256 中文激活版
 化学分析数据处理套件 Mestrelab MestReNova v14.2.0.26256 中文激活版
化学分析数据处理套件 Mestrelab MestReNova v14.2.0.26256 中文激活版